 TuttoChimica.it
TuttoChimica.it
Chimica, laboratorio e dintorni...

Analisi della sequenza di proteine
Sequenziare una proteina
L'analisi della sequenza primaria di proteine avviene frammentando un amminoacido alla volta a partire dall'estremità N-terminale. Staccando un amminoacido alla volta con un opportuno reattivo, e riconoscendo l'amminoacido con un opportuno metodo di rivelazione, potremmo ricostruire la sequenza.
1) La prima cosa che dobbiamo considerare è che non tutte le proteine sono costituite da una singola subunità, e che a volte le subunità proteiche possono essere tra loro legate da ponti disolfuro (come ne lcaso dell'insulina). Pertanto la prima cosa da farsi è una denaturazione e un taglio dei ponti disolfuro tramite ossidazione o riduzione.
Per denaturare una proteina bastano soluzioni a pH estremi, ma non tali da idrolizzarla: solitamente si utilizza una soluzione 8M di urea, oppure una soluzione 6M di cloruro di guanidinio. In alcuni casi sono invece sufficienti alte concentrazioni saline per denaturare la proteina.
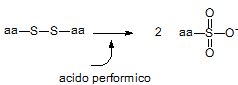
Per rompere i ponti disolfuro con ossidazione si può utilizzare acido performico, ottenendo due residui di acido cistico:
Per rompere i ponti disolfuro tramite riduzione si può utilizzare ditiotreitolo o mercaptoetanolo, seguita da ossidazione con iodio acetato per evitare che i gruppi tiolici formatisi reagiscano riformando il ponte disolfuro:

2) Dobbiamo considerare che non è possibile studiare sequenza di amminoacidi (ma questo è un problema che vale anche per il DNA) superiori ai 40-60 monomeri. Pertanto si dovrà precedentemente frammentare la sequenza proteica tramite enzimi specifici che tagliano la sequenza in corrispondenza di zone note (per esempio la tripsina taglia in corrispondenza di Arginine o Lisine), o metodi chimici che tagliano in punti ben precisi. Una volta spezzato il filamento, i frammenti potranno essere sequenziati.
3) A questo punto, ottenuti i singoli frammenti, li sequenziamo, cioè ne studiamo la sequenza di amminoacidi. Il reattivo che si può utilizzare è il reattivo di Edman, il fenilisotiocianato:
![]()
In ambiente alcalino, il fenilisotiocianato si lega all'estremità N-terminale (all'NH2 terminale). A questo punto si varia il pH ad acido e si tratta la miscela con acido trifluoroacetico, il quale stacca il derivato che comprende il fenilisotiocianato legato all'amminoacido terminale. Questo derivato, chiamato PTH (feniltioinantoina) è in seguito studiabile con metodi di rivelazione per determinare che amminoacido sia. Una volta staccato il primo amminoacido, ciclicamente vengono ripetute le operazioni per staccare gli amminoacidi seguenti.
Questa operazione viene effettuata in appositi strumenti, detti sequenziatori, che permettono di fissare la proteina ad un supporto, consentendo di separarla dall'amminoacido che si stacca volta per volta, ad ogni ciclo di trattamento.
Un sequenziatore diffuso è il sequenziatore a coppa rotante, dotato di una specie di bicchiere centrale che ruota continuamente: la proteina si disporrà lungo la parete, il solvente viene fatto passare sulle pareti, dove avvengono le reazioni, e l'amminoacido quando si stacca viene aspirato via dalla parte centrale dello strumento, mentre la proteina resta adesa alla parete rotante.
Un altro sequenziatore utilizzato attualmente prevede l'utilizzo di un filtro di fibre di vetro che consente di immobilizzare la proteina e di convogliarle sopra i vari reagenti.
4) Il passaggio successivo consiste nel riconoscimento degli amminoacidi staccati. Questo può essere effettuato tramite HPLC. Se nel corso dell'operazione di sequenziamento si sono staccati due amminoacidi, si otterranno in tal caso due picchi, di cui quello maggiore dovuto all'amminoacido in analisi al momento. All'aumentare della posizione dell'amminoacido nella sequenza del frammento, la confusione in termini di picchi aumenta: per questo motivo non si possono analizzare sequenza superiori ai 50-60 monomeri contemporaneamente. Mettendo i vari PTH in HPLC e confrontandoli coi picchi di standard noti, si può risalire all'amminoacido. Da notare che il fenilisotiocianato ben si presta all'uso in HPLC per via dell'anello benzenico rilevabile dal rivelatore UV dell'HPLC.
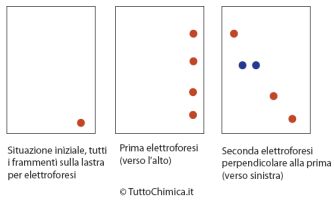
5) L'ultimo passaggio consiste nel riunire le informazioni in possesso sui vari frammenti e sequenze ottenute e ricostruire la proteina. E' interessante notare che il taglio dei ponti disolfuro viene effettuato inizialmente pur non sapendo se tali ponti ci siano o meno, e in che frammento eventualmente si trovino. Pertanto si dovrà effettuare un ulteriore studio per valutarne o meno la presenza. Viene effettuato in questo modo: si idrolizza la proteina, senza tagliarne i ponti dosolfuro, nei singoli frammenti (come si è fatto al punto 2), e si fa una elettroforesi, tecnica che separa i frammenti, in base alla loro carica, su un apposito gel in cui è fatta passare corrente. L'elettroforesi effettuata è bidimensionale: prima si effettua l'eletroforesi con la lastra in posizione nella cella elettroforetica; i vari frammenti si separeranno in base alle relative cariche; quindi si tratta la lastra con acido performico, che taglia i ponti disolfuro, e si capovolge di 90°la lastra. A questo punto si rieffettua una seconda elettroforesi, che sarà quindi perpendicolare alla prima. Per i frammenti che non contengono ponti disolfuro nulla cambia, e continuano a muoversi con la stessa velocità della prima elettorforesi: il frammento che invece contiene ponte disolfuro si separerà in due macchie, dovute alla differenza di carica tra i due frammenti tenuti assieme dal ponte disolfuro.
Categorie Articoli e Appunti
• Chimica Generale
•
Chimica Analitica
•
Chimica Inorganica
•
Chimica Organica
•
Biochimica
•
Biologia
•
Genetica